尤度や情報量規準を使ったグラフィカルラッソ(glasso)に対するクロスバリデーション
グラフィカルラッソのハイパーパラメータはクロスバリデーション(CV)でも決めることができます。
この辺は、例えば井出先生がサラッと言及1してたり、論文を漁ると普通に「CVで決めました」などと書かれていますが、日本語で具体的にどうやっているかを書いているものが見当たらなかったので紹介します。また、これを通してCVによるハイパーパラメータの決定はglassoにおいてはあまり実用的でないかもしれないということをお伝えします。なお、ガウシアングラフィカルモデルやグラフィカルラッソに関する詳しい説明はしません。
グラフィカルラッソ(glasso)とは
ガウシアングラフィカルモデル(GGM)の対数尤度関数にL1ノルムを加えた定式化と、それを高速に解くアルゴリズムを指します。\(n\)個のデータが得られた時の\(p\)変量正規分布の対数尤度関数は次のように書けます。
$$\frac{n}{2}(-p\text{log}(2\pi) + \text{log}|\Omega| – \text{tr}(\Omega S))$$
ここで、\(\Omega\)は精度行列(共分散行列の逆行列)であり、この問題における決定変数(推定するパラメータ)で、\(S\)は標本共分散行列を表します。2つ目の\(\text{log}\)の中にある\(|\cdot|\)は行列式を表し、\(\text{tr}(\cdot)\)は対角成分の和を計算する操作です。この関数から定数項や係数を(最適化に影響のない範囲で)取り除いて\(-1\)をかけ、L1ノルムを加えた以下の損失関数をブロック座標降下法(Blockwise coordinate descent)で反復的に解くのがglassoです。
$$-\text{log}|\Omega| + \text{tr}(\Omega S) + \lambda\|\Omega\|_1$$
\(\lambda\)はどれだけスパースな推定をするかを調節する罰則パラメータで、\(\|\Omega\|_1\)は行列の各要素の絶対値の和を表します。
ちなみに、なぜ精度行列のスパース推定がスパースなグラフ推定になるのかを説明すると、多変量正規分布において\(x_i\)以外の変数\(x_{-i}\)によって条件つけられた\(x_i\)の分布(条件付き分布)は次のようにかけます。
$$p(x_i|x_{-i})=\mathcal{N}(-\frac{1}{\omega_{i,i}^2}\sum_{j \neq i}^p{\omega_{i,j}x_j}, \frac{1}{\omega_{i,i}^2})$$
\(\omega_{i,j}\)は精度行列の\((i,j)\)成分です。この式から、\(\omega_{i,j}=0\)であれば、\(x_i\)の条件付き分布に\(x_j\)の値は無関係、つまり条件付き独立であるということができます。これにより、\(\omega_{i,j}=0\)なら\(x_i, x_j\)は条件付き独立なのでエッジなし、\(\omega_{i,j}\neq0\)なら関係ありということでエッジを引く、とすることでスパースなグラフを構築することができます。
CV内での評価に使う関数
回帰であれば予測誤差の二乗和などが使われると思いますが、グラフィカルラッソの場合、対数尤度をベースにした関数を使用します。こちらがよく使われる関数です2。
| 関数名 | 式 |
| -最大対数尤度:\(\ell(\hat{\Omega})\) | \(-\frac{n}{2}(-p\text{log}(2\pi) + \text{log}|\hat{\Omega}| – \text{tr}(\hat{\Omega} S))\) |
| 赤池情報量規準(AIC) | \(-2\ell(\hat{\Omega}) +2|\boldsymbol{E}|\) |
| 拡張ベイズ情報量規準(EBIC):\(\text{EBIC}(\boldsymbol{E}, \gamma)\) | \(-2\ell(\hat{\Omega}) +|\boldsymbol{E}|\text{log}n + 4|\boldsymbol{E}|\gamma\text{log}p\) |
| ベイズ情報量規準(BIC) | \(\text{EBIC}(\boldsymbol{E}, \gamma=0)\) |
\(|\boldsymbol{E}|\):非対角成分の非ゼロエッジ数/2(推定されたグラフのエッジ数)
なお、\(n\)は検証データのサンプルサイズです。
実験
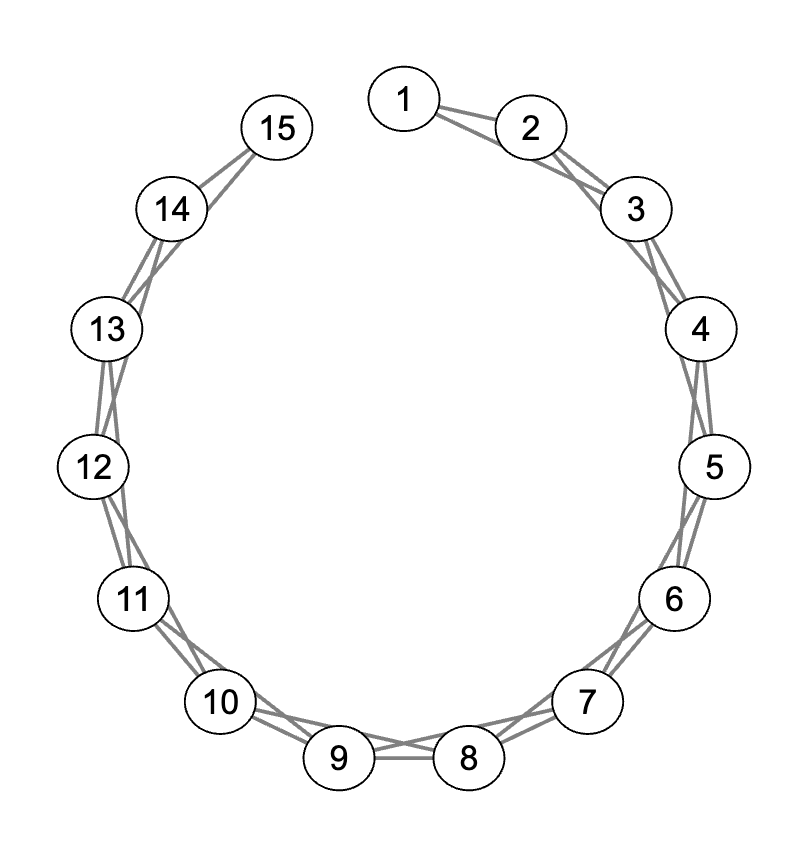
こちらの図のグラフ構造になるように真の精度行列を設計し、その逆行列である真の共分散行列を使った多変量正規分布からデータをサンプリングします。これは1つ隣と2つ隣のノード同士は相関を持つグラフで(1番目と最後は繋がない)、1つ隣の方が2つ隣よりも強い相関を持つように生成してます。特徴量(ノード)数は15、サンプルサイズは150, 1500の二通りでやってみます。
generate_data = function(p, n) {
true_omega = diag(p)
for (i in 2:p) {
true_omega[i, i-1] = 0.5
true_omega[i-1, i] = 0.5
if (i > 2) {
true_omega[i, i-2] = 0.25
true_omega[i-2, i] = 0.25
}
}
true_s = solve(true_omega)
set.seed(0)
data = rmvnorm(n, mean=rep(0,p), sigma=true_s)
data_cov = cov(data)
true_graph = ifelse(true_omega!=0 & row(true_omega)!=col(true_omega),1,0)
result = list(
data = data,
cov = data_cov,
true_cov = true_s,
true_omega = true_omega,
true_graph = true_graph
)
return(result)
}このデータを使い、5分割CVで\(\lambda\)を決定する際、使用する評価関数によって結果がどうなるのかを比較していきます。なお、EBICでは\(\gamma=0.5\)としました。
N=150の場合
data_info = generate_data(p=15, n=150)
set.seed(0)
n_folds = 5
data_cv = data_info$data |> vfold_cv(v=n_folds)
loglik = c()
aic = c()
bic = c()
ebic = c()
calc_loglik = function(omega, s, n) {
p = ncol(omega)
return(n/2 * (-p*log(2*pi) + log(det(omega)) - sum(diag(omega %*% s))))
}
calc_E = function(omega) {
return(sum(omega[lower.tri(omega)] != 0))
}
for (i in 1:n_folds) {
train = analysis(data_cv$splits[[i]])
val = assessment(data_cv$splits[[i]])
cov_train = cov(train)
cov_val = cov(val)
n_val = nrow(val)
models = apply(t(lambda_range), 2, function(lam) glasso(s=cov_train, rho=lam))
loglik_fold = mapply(function(model) -1*calc_loglik(model$wi, cov_val, n_val), models)
aic_fold = mapply(function(model) -2*calc_loglik(model$wi, cov_val, n_val) + 2*calc_E(model$wi), models)
bic_fold = mapply(function(model) -2*calc_loglik(model$wi, cov_val, n_val) + calc_E(model$wi)*log(n_val), models)
ebic_fold = mapply(function(model) -2*calc_loglik(model$wi, cov_val, n_val) + calc_E(model$wi)*log(n_val) + 4*calc_E(model$wi)*0.5*log(ncol(cov_val)), models)
loglik = cbind(loglik, loglik_fold)
aic = cbind(aic, aic_fold)
bic = cbind(bic, bic_fold)
ebic = cbind(ebic, ebic_fold)
}
loglik_mean = loglik |> apply(1, mean)
aic_mean = aic |> apply(1, mean)
bic_mean = bic |> apply(1, mean)
ebic_mean = ebic |> apply(1, mean)長いので可視化部分のコードは端折りますが、こちらが\(\lambda\)に対応する評価関数値を評価関数別にプロットしたものです。縦線はそれぞれの評価関数における最小値(=選択する\(\lambda\))です。
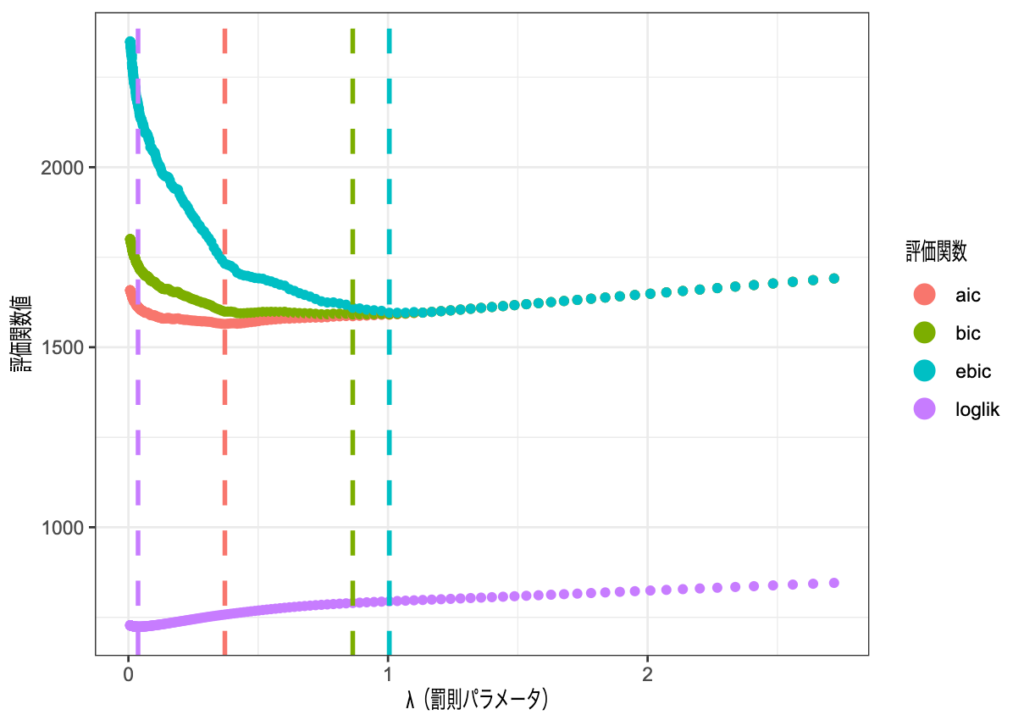
これは理論通りな結果で、情報量規準は最大対数尤度にモデルの自由度に関する罰則を加えたものになっているので、選択される罰則パラメータも大きくなりやすいです。さらに、EBICはBICの罰則をより強めたものなので、BICで選択される\(\lambda\)より大きくなっているのも納得です。
次に、それぞれの最適\(\lambda\)で学習したモデルによるグラフを可視化します。
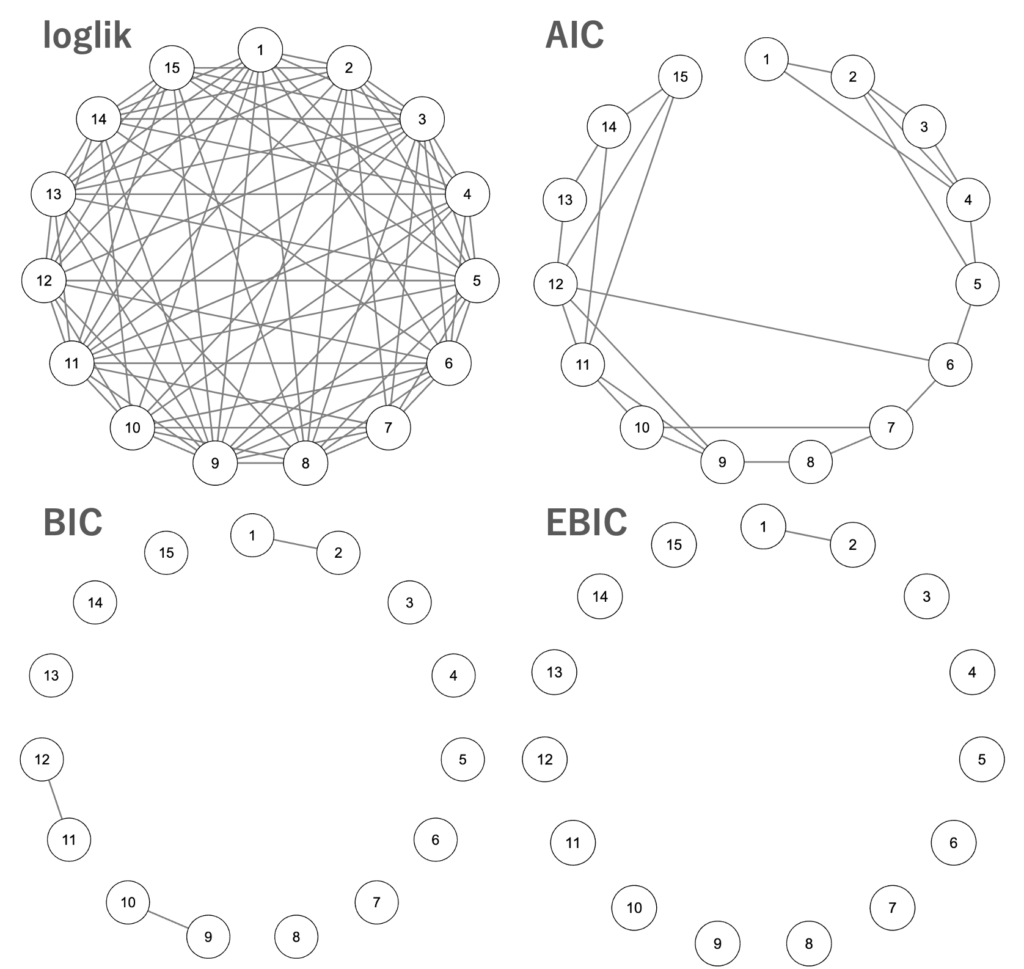
loglikは見るに堪えないグラフですね。BIC, EBICは慎重に選びすぎてもうちょっと情報が欲しいところです。このデータではAICが最も使いやすそうです。また、これは評価関数ではなくglassoのいいところですが、ちゃんと1つ隣のノードとのエッジが優先されて結ばれていることがわかります。
N=1500の場合
図とそれらに対するコメントだけ簡潔に書きます。
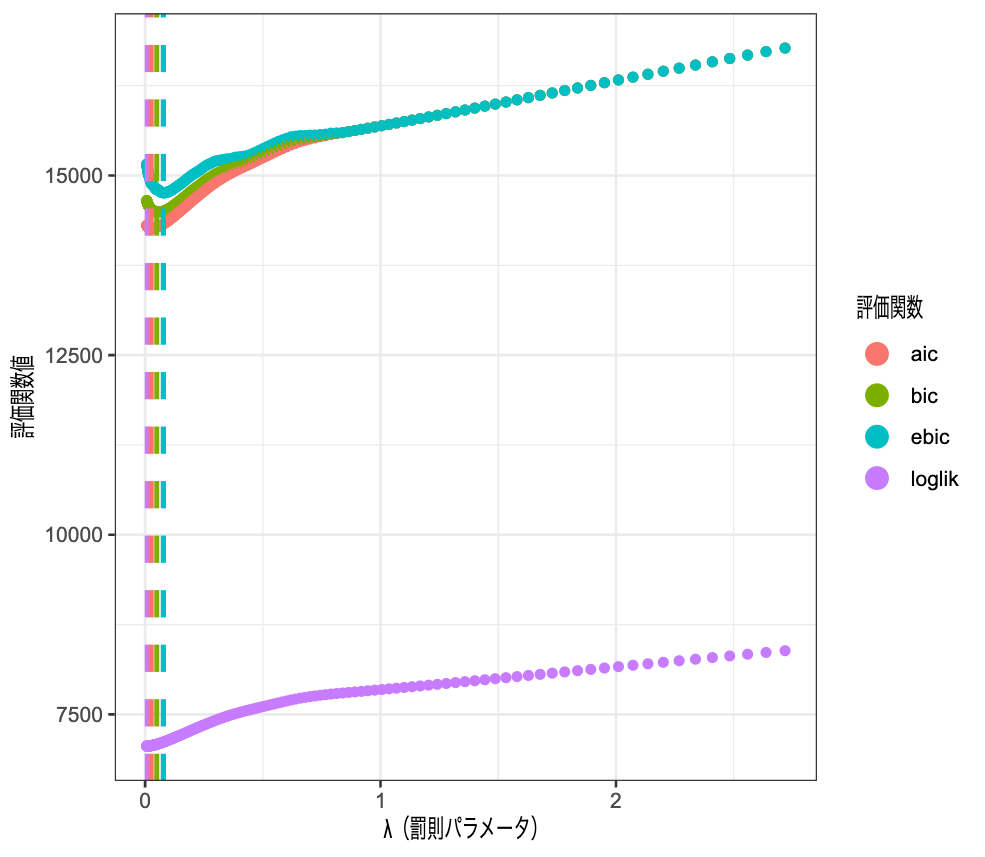
データが増えたおかげで、どの基準でもより小さい罰則で済むようになりました。このような場合、\(\lambda\)をさらに細かく、小さい値のところで取り直すのが適切だと思います。最小となる\(\lambda\)の大小関係は先ほどと同様です。
次に推定されたグラフです。
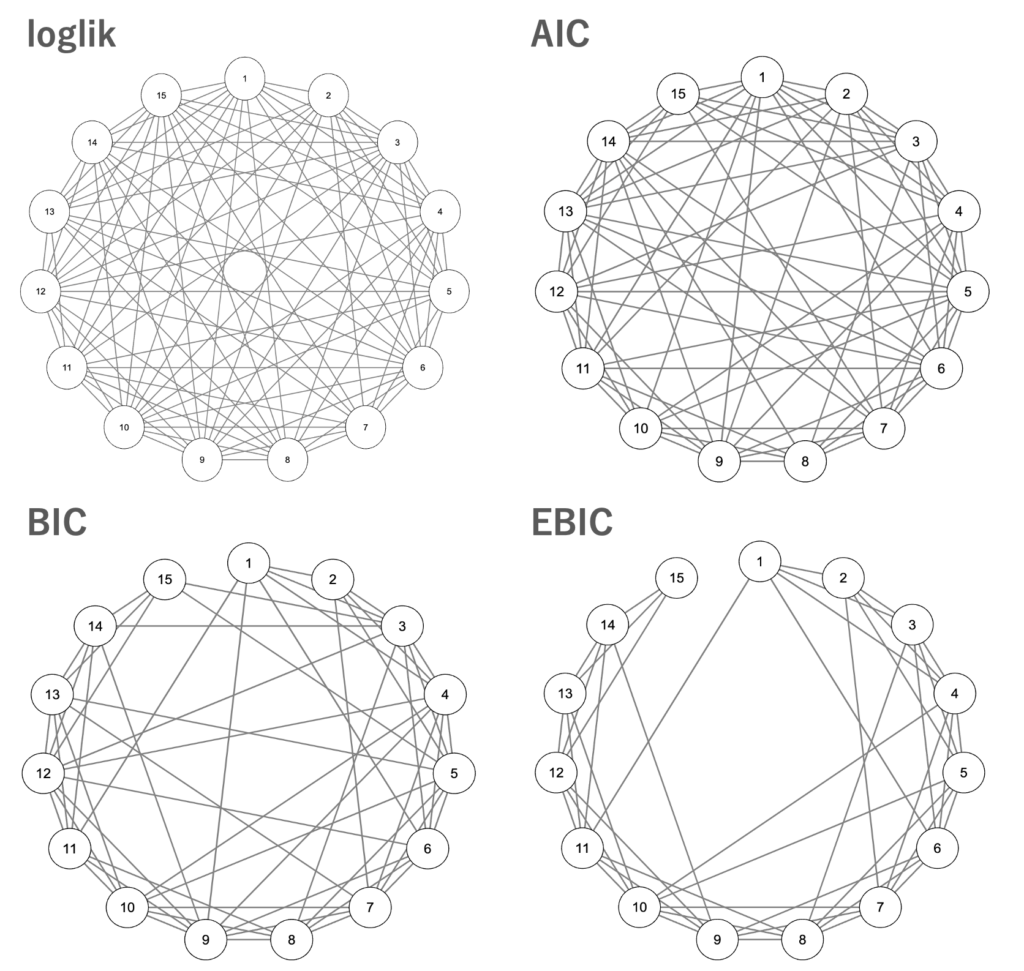
loglikはあり変わらずのグラフで、AICもかなり複雑になってしまいました。BICもかなり複雑ですが、EBICはかなりいい感じに推定できています。
結果を通して
サンプルサイズが異なるだけで選択される\(\lambda\)がかなり異なることがわかりました。また、それにより\(\lambda\)の探索範囲もデータによって調節する必要があります。
また、loglikを評価関数に使うとかなり複雑なグラフが推定されてしまい、真のグラフから大きく乖離しているため実用上好ましくありません。
サンプルサイズが大きくなると、各評価関数における最大対数尤度部分の影響が強くなってしまい、結果としてloglikと似たような選択をする(=小さい罰則を選ぶようになる)と考えられます。
glassoは真のグラフを推定するため、というより、EDAの一環としてデータの理解や仮説構築のために使うことが多いのではないでしょうか。そうなると、このような決定方法より、最終的には自分が解釈しやすいレベルのグラフを生成するように\(\lambda\)を調節する、というのが実用上はいいのかもしれません。
参考
- 井出剛, 依存関係にスパース性を入れる(https://ide-research.net/papers/2016_Iwanami_Ide.pdf) ↩︎
- Foygel, R., & Drton, M. (2010).
Extended Bayesian information criteria for Gaussian graphical models.
Advances in neural information processing systems, 23. ↩︎